 2025-04-10
2025-04-10
 0
0
CRISPR筛选后的高通量测序分析是获得关键功能基因的重要的步骤,通过分析实验组和对照组之间sgRNA的富集或者丢失情况,可以清晰了解哪些基因功能受到干预后可以产生研究者感兴趣的表型。研美生物的CRISPR文库筛选分析报告主要是基于MAGeCK分析获得结果进行了可视化以及初步的解读,但是对于初次接触CRISPR筛选甚至对高通量测序本身不熟悉的人来说,要想充分理解报告的内容通常会遇到很多疑问。今天,我们针对主要的一些问题为大家解答一下,以期大家更好地理解报告的内容,提高工作效率。
1. 一个样本二代测序需要测多少数据量合适?
为了充分覆盖CRISPR文库的每个sgRNA,我们推荐测序深度为300-500X。比如:您的文库包含了10,000条sgRNA,如果您计划测500X的数据量的话,最终需要测10,000 x 500 = 5,000,000条即5M reads。双端测序的情况下,最终的碱基数就是5M x 300 nt = 1500M nt,即大约1.5G的数据量。考虑到还存在数据预处理和去接头的后续操作会降低实际Mapping至sgRNA的数据量,一般建议10,000条sgRNA测2G数据量。
2. Mapping率比较低,筛选的结果可信吗?
Mapping率是指测序获得的reads比对到CRISPR文库中sgRNA的比例。CRISPR筛选NGS文库的制备是基于PCR反应的靶向建库。由于可能存在基因组DNA浓度低,阳性基因组DNA占比低和过度扩增导致的副反应,常会导致PCR产物中富集非特异性的产物,从而导致mapping率比较低。一般来说,如果mapping率大于50%的情况下测序深度大于300x,数据可以用于后续分析,Mapping低于50%则可能会导致数据分析失真。
3. 什么是负向筛选(negative screen),什么是正向筛选(positive screen)?
neg代表的是负向筛选:以药物筛选为例,其意义是在筛选压力下导入文库sgRNA后,细胞对压力表现出更加敏感,促进细胞死亡。pos则代表正向筛选:以药物筛选为例,其意义是在筛选压力下导入文库sgRNA后,细胞更加耐受药物压力,抑制细胞死亡。简言之,neg基因代表通过你施加的某种压力后,在最终的细胞群体中,靶向该基因的sgRNA细胞与对照比变少了,正向则相反。报告文件夹中的rra_x.gene_summary中提供了按照RRA score排序后的筛选结果,示例图片展示了Top10基因的位置。
4. 为什么负向筛选的存在LFC正值?而正向筛选结果中存在LFC负值?
我们会发现有时候负向筛选的LFC为正值,相反有时候个别正向基因的LFC则为负值。这主要是neg.score和pos.score是按照RRA算法计算得到的打分,它通过整合多个sgRNA的排名信息,评估基因在筛选实验中的全局重要性,适用于两组(如处理组vs对照组)的比较分析。LFC则是通过同一基因对应的所有sgRNA的log2FC值进行汇总,生成基因水平的neg.lfc/pos.lfc。有时候个别sgRNA的LFC偏差非常大,这会导致整体水平上的LFC的偏倚。例如在负向筛选中,A基因有3个sgRNA,log2FC[sgRNA1]为-1.2,log2FC[sgRNA2]为-1.5,log2FC[sgRNA3]为3.6,从统计学的角度两条负向good sgRNA一条正向good sgRNA会被归为neg基因,但其LFC倍数为(3.6-1.2-1.5)/3=0.3。正向筛选出现LFC负值也是类似的情况,我们可以在rra_x.sgRNA_summary文件中找到每一个基因所有对应sgRNA的变化情况。通常,我们可以设定neg的LFC为-1或者pos的LFC为1过滤掉此类存在冲突的基因。
5. 按照RRA score排序筛选基因合适还是按照LFC与p值联合筛选基因合适?该如何挑选目的基因?
无论是从RRA score还是从LFC的角度来对筛选文库基因排序都是可行的,需结合常见的显著性标准:全库FDR小于0.05,亚库FDR小于0.1。如果出现FDR均大于0.1的情况,可以使用p值进行筛选。
值得注意的是,不管用哪个参数来筛选目的基因,good sgRNA的参考也是非常重要的。一般来说,3条sgRNA需要有两条good sgRNA。对筛选出的TOP基因进行原始数据分析时,一个有效的筛选结果应表现为同一基因的多个sgRNA变化趋势保持一致性。如果筛选结果中仅发现单一sgRNA表现出显著的偏差,这可能意味着该基因的筛选结果的可靠性存疑。
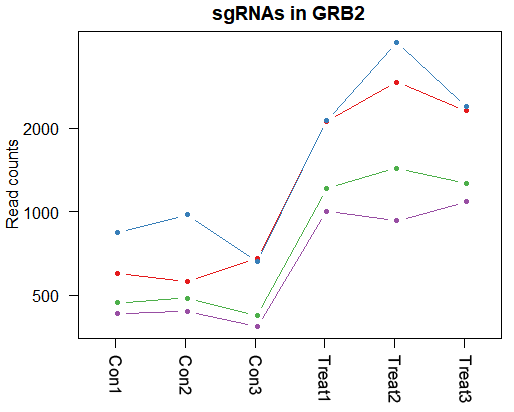
图1. good sgRNA在对照组和处理组中的分布
最后,通过检索相关文献,寻找在细胞系、筛选药物、筛选压力和时间等方面与本实验相似的研究。通过对比这些文献中的筛选结果与本实验的结果,我们可以评估本实验筛选出的TOP基因与文献中报道的TOP基因的一致性。如果本实验的筛选结果在很大程度上与文献中的阳性对照基因相吻合,这将表明本实验的筛选结果具有较高的可靠性和质量。
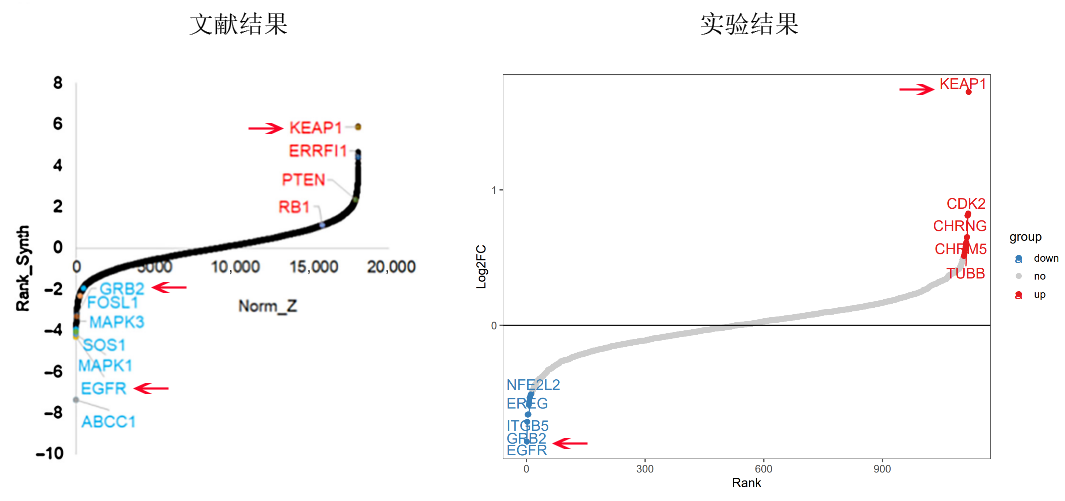 图2. 实验结果与文献阳性基因比较
图2. 实验结果与文献阳性基因比较
6. 测序结果显示样本丢失了很多sgRNA怎么办?
经过压力筛选后的文库,由于某些sgRNA的导入后细胞对压力更加敏感,这部分细胞会最早开始死亡,因此这一部分的细胞群体会慢慢变小,甚至完全消失。筛选压力越大,丢失sgRNA可能就越多。FACS筛选中通常因为分选细胞群比例的差异(如Top 5%/10%/20%),因比例越低会丢失的越多。这些都是正常现象。但是如果药物筛选压力不大,比如IC50压力下筛选,仍然丢失了过多的sgRNA则应考虑是否是筛选实验过程中传代丢失的。应保证每次传代的细胞覆盖文库500x以上,对于全库对照是300x以上,以避免不必要的丢失影响最终实验的准确性。此外,对照组(NC,input)不应该丢失较多sgRNA,应保证95%的sgRNA都不丢失。
7. 基于流式分选(FACS)结果如何理解?
在基于FACS的CRISPR筛选中,如果以抗原A分选的细胞群(Top 10%或Bot 10%)与未分选细胞群(Input)比较时,Top组或Bot组neg筛选实际意义有限。因为筛选压力大,sgRNA可能随机性丢失,当分选组sgRNA丢失时与input相比在neg这个方向随机富集。有一定参考意义的是,靶向抗原A的sgRNA应该在Bot组neg中富集,而在Top组neg中减少。pos筛选基因则表示导入该基因sgRNA后,A的表达降低(Bot,A-low)或者升高(Top,A-high)的细胞相对于input变多,这样可以分别筛选获得A的增强和抑制调控基因。有时候,研究者还会使用A-low与A-high细胞群进行比较。此时,neg筛选基因则表示敲除该基因后,A的表达降低了(因为neg组sgRNA丢失率的问题,neg组数据可能意义有限),pos筛选基因则表示A的表达升高了。因此,不同的比较方式其意义是不一样的,研究者应当注意这样的不同。
8. 样本的重复性不好?
报告中提供了PCA分析和样本聚类分析的热图结果,对于相同处理的组内重复样本,聚类分支上应表现为距离临近。有时候由于实验的批次不同导致个别样别离群严重,可以考虑去除离群样本,尽量获得重复性更好的样本进行分析,以获得更加趋近真实的结果。
9. 有多个重复的样本,进行多组一对一的分析好还是多样本一起分析比较好?
我们建议将生物学重复一起分析以最大限度展示筛选结果的可重复性。一对一的分析存在一定的偶然性结果。生物学重复可以一定程度上减少随机性结果带来的偏差。



